Ein Steckenpferd unserer Höhlengruppe ist die Fledermausforschung, die vor allem Dr. Christian Zilch vorantreibt. Die Höhlengruppe berichtet hier über das Metagenom-Projekt, bei dem alle tatkräftig mit anpacken.
Das Projekt
Christian Zilch ist nicht nur Mitglied der Höhlengruppe, sondern auch Initiator des Metagenom-Projektes (detaillierte Infos dazu gibt es hier).
Um unkompliziert an Fledermausproben zu kommen, hat sich die Gruppe den Fangaktionen von Johannes Mohr angeschlossen – ein Fledermaus-Veteran in Bayern, der seit über 20 Jahren Forschung an den faszinierenden Flug-Säugetieren betreibt. Das Team kann

Die Fangnetze sind aufgspannt. Foto: C. Zilch
sich auf seinen großen Erfahrungsschatz bei der Organisation und Durchführung derartiger Expeditionen verlassen. Er kennt – wie auch das Leitungsteam der Höhlengruppe, Jutta und Stefan Uhl, – jede Höhle im Umkreis von Forchheim und der fränkischen Schweiz und hat bereits unzählige Fangaktionen begleitet. Zu jedem Tier und jeder Pflanze, die einem auf dem Weg begegnen, kann Johannes eine kleine Geschichte erzählen.
Die Vorbereitungen
Das Treffen der Fledermausfänger findet eine Stunde vor Sonnenuntergang an einem lauen Herbstabend in Laufweite zu einer Höhle in der fränkischen Schweiz statt. Etwa ein Dutzend Helfer, Fledermausforscher, Biologen, Veterinärmediziner und Mitglieder der Höhlengruppe schleppen umfangreiches Material für das mobile Labor bergauf zum Höhleneingang. In einem der Nebengewölbe werden Tische und Bänke, Beleuchtung, Laborutensilien und Vermessungsgeräte aufgebaut. Feine Fangnetze, so groß wie Volleyball-Netze, werden vor dem Höhleneingang aufgespannt.
So gehen die Fledermäuse ins Netz
Dann teilt sich das Team in mehrere Gruppen auf. Die einen harren nach dem Aufbau neben den Fangnetzen aus. Sie sind mit Batrecordern ausgerüstet, um die Ultraschall-Laute der nachtaktiven Tiere für das menschliche Ohr hörbar zu machen. Hat sich eine Fledermaus im Netz verfangen, ruft Sie besonders laut. Dabei hat jede Art ein typisches Rufspektrum, um sich im dunklen Raum zu orientieren und auf Beutejagt zu gehen.
Anhand des spezifischen Ultraschall-Spektrums lassen sich die einzelnen Fledermaus-Arten auseinanderhalten. In Bayern sind mehr als 20 verschiedene Arten heimisch, von den kleinen Fransen-, Bart-, Nymphen- und Mückenfledermäusen bis zu den großen Mausohren, Abendseglern oder Hufeisennasen, die es in ganz Deutschland nur noch sehr selten gibt.
Hat sich ein Fledertier in den feinen Maschen verfangen, wird es behutsam befreit. In einem sterilen Stoffsäckchen an eine Art Wäscheleine aufgehängt beruhigen sich die Tiere und kühlen ihre Körpertemperatur wieder etwas herunter, bevor sie sich der Beprobung unterziehen müssen.
Das unglaubliche Immunsystem der Fledertiere
Fledermäuse verbrennen für ihre geringe Größe enorme Mengen an Energie, um den aktiven Flug zu unterstützen. Die Stoffwechselrate ist dabei um das 15-Fache höher als im Ruhezustand (zum Vergleich: beim Menschen ist es nur um das 2-3-Fache). Durch die hohe metabolische Aktivität entstehen ständig freie Radikale, die die Zellen schädigen können. Fledermäuse besitzen jedoch einen effektiven Mechanismus zum Abbau der toxischen Metaboliten. Zusätzlich haben die Tiere im Laufe der Evolution besonders kompetente Abwehrkräfte entwickelt. Ihr Immunsystem produziert dabei ständig Interferone – zentrale Botenstoffe der viralen Immunabwehr. Im Gegensatz zum Menschen befindet sich das Immunsystem der Fledermäuse quasi in ständigen Alarmzustand. Dadurch sind sie in der Lage, gefährliche Pathogene besser in Schach zu halten. Das bedeutet aber auch, dass gerade die besonders robusten Viren, die in Fledermaus-Populationen überleben, ein leichteres Spiel haben, wenn sie auf andere Spezies mit weniger ausgeprägten Abwehrkräften überspringen.
Die Fledermäuse werden bestimmt
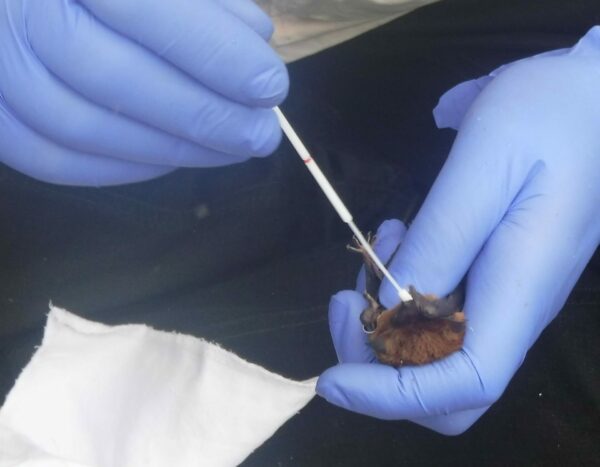
Probentnahme. Foto: C. Zilch
Ist ein Tier gefangen, kommt das zweite Team zum Einsatz: Die Bestimmung der gefangenen Art, des Geschlechts und des ungefähren Alters. Dabei werden die Tiere vorsichtig gewogen, vermessen und auf Verletzungen und Parasiten untersucht. Bei einigen Tieren findet eine Beringung statt, um die Individuen der heimischen Fledermaus-Populationen und deren Verbreitungsregionen zu verfolgen.
Jetzt beginnt der für die Tiere unangenehme Teil der wissenschaftlichen Arbeit: Von jeder Fledermaus werden verschiedene Proben entnommen, Rachen- und Analabstriche, Kotpellets werden aus den Säckchen geborgen. Alle Proben müssen mit einem Code versehen und exakt protokolliert werden, bevor sie schockgefroren werden.
Der Weg der Proben
Sämtliche Proben werden noch in der gleichen Nacht in ein Labor in Forchheim gebracht, um sie bei -80°C in einem Ultratiefkühlschrank zu lagern. Ähnliche Kühlschränke werden auch zur Aufbewahrung von mRNA-Impfstoffen eingesetzt, da die Nukleinsäuren extrem temperaturempfindlich sind – nicht nur die in den Impfstoffen, sondern auch die in den Fledermaus-Proben. Die Kühlschränke dienen ausschließlich der Lagerung der Umweltproben, bevor sie in ein virologisches Hochsicherheits-Labor am Robert-Koch-Institut (RKI) in Berlin versandt werden.
Und was macht das Probensammelteam?
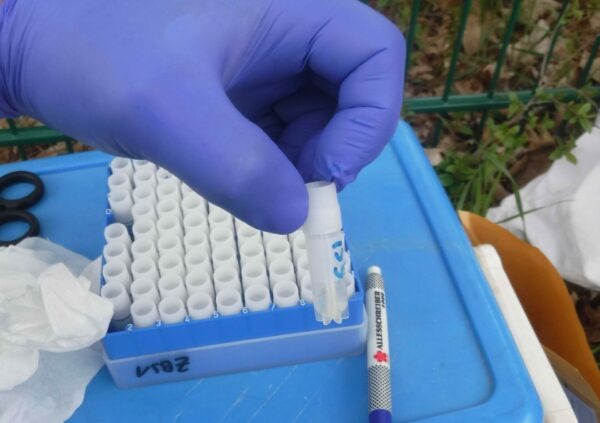
Jede Probe muss codiert werden. Foto: C. Zilch
Weit nach Mitternacht, wenn die letzten Fledermäuse beprobt und die Fangnetze wieder eingerollt sind, bereitet sich das Team auf das Finale vor: Alle Fledermäuse werden aus den Stoffsäckchen wieder in die Freiheit entlassen. Einige der Tiere schlafen nach der anstrengenden Prozedur bereits, die Körpertemperatur ist deutlich vermindert. Sie müssen sich erst aufwärmen, bewegen sich langsam und machen sich mit der Umgebung vertraut, bevor sie schließlich in das Dunkle der Nacht abheben. Andere Tiere flattern gleich nach dem Öffnen des Stoffsäckchens in die sehnsüchtig erwartete Freiheit.
Die Aktion war erfolgreich. Mehrere Dutzend Fledermäuse wurden eingefangen und beprobt. Jetzt heißt es nur noch, die Proben unbeschadet zu den Virolog*innen nach Berlin zu bringen und mit Spannung auf die Ergebnisse der Metagenom-Analyse zu warten.
Bis die Resultate vorliegen, werden die Fledermäuse schon lange aus ihrem nächsten Winterschlaf erwacht sein.
Im Auftrag der Virenforschung
Die Projekpartner*innen am Zentrum für Biologische Gefahren und Spezielle Pathogene am RKI widmen sich seit Jahren der Virenforschung. Ein hochmodernes Werkzeug bietet die so genannte Metagenomanalyse. Dabei isoliert man keinen „lebenden“ (replikationsfähigen) Erreger, sondern lediglich deren Nukleinsäuren (DNA und RNA) aus der gesamten Probe. Jeder kann sich vorstellen, dass sich bspw. in einer Kotprobe unzählige Bakterien, Pilze, Parasiten, Viren und teilweise Zellen (Darmepithelzellen) des Wirtes befinden. Ein riesiges Sammelsurium von Nukleinsäure-Fragmenten wird so aus einer einzigen Probe isoliert. Wie findet man da die sprichwörtliche Nadel im Heuhaufen?
So werden neue Viren entdeckt
Als modernste Methode gilt das Next-Generation-Sequencing (NGS). Bei der Vorbereitung der Probe wird an jedes der Millionen von Nukleinsäure-Stücken ein spezieller Adapter angefügt. Man kann sich das vorstellen wie Anker an jedem Ende eines Seilstrangs. Das Sequenzier-Gerät liest jetzt die Buchstabenreihenfolge des genetischen Codes jedes dieser Millionen von kleinen Stücken, während eine spezielle Software sie wieder zu kompletten Genomen zusammenfügt.
Vereinfacht könnte man sich das Genom eines „CORONAVIRUS“ vorstellen, das in verschiedene Fragmente zerteilt ist. Die Sequenzier-Maschine liest grob vereinfacht folgende drei Teilstücke: CORONA – ONAVIR – VIRUS
Aus diesen drei Teilstücken, die in der Mitte überlappen, kann die Software das Gesamtwort „CORONAVIRUS“ zusammenfügen.
Ähnlich funktioniert das mit den 4 Buchstaben des genetischen Codes, der auf Genom-Fragmenten mit einer Länge von mehreren hundert bis tausend Buchstaben aneinandergereiht ist. Die Sequenzier-Maschine liest die Buchstabenreihenfolge während die Metagenom-Software die überlappenden Stücke wieder zu kompletten Genomen zusammenfügt. Diese werden anschließend mit Datenbanken abgeglichen, in der die Genome sämtlicher bekannter Organismen der Welt gespeichert sind (die umfassendste Datenbank ist die des NCBI, „National Center for Biotechnology Information“).
Auf diese Weise kann schlussendlich die gesamte Vielfalt von Organismen in einer Probe bestimmt werden. Selbst neue Pathogene, bspw. ein neuartiges Virus, enthalten genetische Ähnlichkeiten mit bekannten Viren und können so taxiert, also einem bekannten Organismus zugeordnet werden. Unterschiede zwischen Virussequenzen haben es Forschern ermöglicht, einen viralen Stammbaum zu erstellen, der es ermöglicht, die Verwandtschaftsbeziehungen zu ergründen und das pandemische Potential neu entdeckter Viren einzuschätzen.
Der Sinn hinter dem Metagenom-Projekt
In dem Metagenom-Projekt arbeiten Dr. Zilch und Höhlengruppe eng mit Artenschützern zusammen und erörtern auch kritische Fragen: Macht es überhaupt Sinn, die Pathogene in heimischen Arten zu bestimmen? Besteht nicht die Gefahr, dass man gefährliche Krankheitserreger von entfernten Höhlen in die Zivilisation einträgt? Schürt man durch die Forschung unnötig Ängste in der Bevölkerung vor Virus-Trägern, die eigentlich unter dem Artenschutz stehen?
“Je mehr wir wissen, desto besser”
Christian Zilch, der sich seit mehreren Jahrzehnten mit der Pathogenforschung und der Entwicklung von Nachweissystemen befasst, sagt dazu: „Der Mensch dringt immer tiefer in die Habitate von Wildtieren ein. Dadurch steigt auch die Gefahr von „Spill-Overn“. Es ist wichtig, die Vielfalt von Pathogenen in der Natur zu bestimmen, um zukünftige Pandemien vorhersagen zu können und Impfstoffe dagegen zu entwickeln. Je mehr wir über die natürlichen Pathogene wissen, desto besser können wir präventive Strategien entwickeln.“
Übertragungsgefahr ist gering
Dabei handelt es sich ausdrücklich nicht um die berüchtigten „Gain-of-Function“ Experimente, die die Übertragbarkeit oder Pathogenität von Viren im Labor erhöhen. Letztere sollten seiner Meinung nach nicht in Labors innerhalb dicht besiedelter Regionen erfolgen, selbst wenn es sich um Hochsicherheits- (BSL-4) Labors handelt. Die Gefahr hingegen, dass Erreger aus der Natur durch deren Sammlung in die Zivilisation gelangen, ist äußerst gering, da nicht nur unter entsprechenden Schutzmaßnahmen gearbeitet wird, der bei üblichen Tier-Mensch Kontakten kaum existiert, sondern weil die Proben steril verpackt und in speziellen Gefäßen tiefgefroren werden.
Eine viel größere Gefahr geht von der Nutzung mit Pathogenen kontaminierter Exkremente als Düngemittel aus. Auch werden die Proben in den Hochsicherheits-Labors durch die Isolation der darin befindlichen Nukleinsäuren lysiert – dadurch werden sämtliche Pathogene deaktiviert.
Gefährliche Viren in Hufeisennasen
Besonders in den Hufeisennasen (Fledermaus-Gattung Rhinolophus) wurden vor allem in bestimmten Regionen Asiens, Südamerikas und Zentralafrikas die bisher gefährlichsten Viren gefunden. Sie bilden quasi ein natürliches Reservoir nicht nur für SARS-ähnliche Viren, sondern auch für Ebola-, Hendra-, Nipah-, Lyssa-, Marburg- und MERS-Viren.
Von den in Europa ansässigen Fledermaus-Populationen geht nicht diese hohe Gefahr aus. Die hiesigen Populationen sind deutlich kleiner und tragen nicht die Vielfalt an gefährlichen Krankheitserregern in sich. Zwar können sie, neben den bekannten Lyssa-Viren (Erreger der Tollwut), wie fast jede Wildtierart auch mit Coronaviren infiziert sein – die sind aber meist mit harmlosen Erkältungsviren verwandt und stellen so keine besondere Gefährdung für den Menschen dar.
Nicht jedes Virus ist auf den Menschen übertragbar
Selbst wenn eines der zahlreichen Fledermaus-Viren entdeckt wird, heißt das noch lange nicht, dass das Virus direkt auf den Menschen übertragbar ist. Meist springen die Viren erst auf so genannten Zwischenwirte wie Ratten, Schleichkatzen, Marderhunde oder Dromedare über, bevor sie auf den Menschen übertragen werden können. Die Zwischenwirte weisen physiologische Merkmale des Atemwegs-, Kreislauf- und Immunsystems auf (besonders die Andockstellen auf den Zielzellen), die dem Menschen ähnlicher sind, als bei Fledermäusen. Die Viren müssen sich über einen längeren Zeitraum sukzessive an ihre neuen Wirte anpassen und benötigen mehrere Anläufe, bis die Grenzen zwischen den Arten überschritten werden können.
Text: Jutta Uhl (nach Vorlage von Dr. Christian Zilch)
Bilder: Christian Zilch











